Clustering
1) 개요
- 유사한 속성들을 갖는 샘플들을 묶어 그룹으로 나누는 것
- unlabed sample을 그룹별로 나누는 비지도 학습의 대표적인 예이다.
2) 유사도 척도 (관측치 간의 거리)
- 거리 측정법은 아래 포스트에 정리돼있다.
k-NN (Nearest Neighbors)
Instance-based learning으로 별도의 모델 생성없이 인접 데이터를 분류 또는 예측에 사용 (비선형) Nearest neighbors 1) 새로운 데이터에서 가장 가까운 거리에 있는 이웃값들의 범위를 의미함 - k-nearest neig
sjpyo.tistory.com
Hierarchical Clustering (계층적 군집화)
1) 개요
- 개체들을 가까운 집단부터 차근차근 묶어나가는 방식
- dendrogram 생성 가능 (이진트리 형태로 계층 군집 시각화)
- cluster 개수를 미리 지정할 필요가 없다.
2) Divisive hierarchical clustering (분할 계층 군집)
- 전체 샘플을 포함하는 하나의 클러스터에서 시작하여 더 작은 클러스터로 반복적으로 나눔
- 각각의 샘플들이 클러스터 안에 할당될 때까지 수행
3) Agglomerative hierarchical clustering (병합 계층 군집)
- 각 샘플이 독립적인 클러스터가 되고 하나의 클러스터가 남을 때까지 가장 가까운 클러스터를 합침
4) 과정
(1) 모든 개체들 사이의 거리에 대한 유사도 행렬 계산
(2) 거리가 인접한 관측치끼리 군집 형성
(3) 유사도 행렬 업데이트
(4) 위의 과정 반복
5) 코드 예시
from sklearn.cluster import AgglomerativeClustering
ac = AgglomerativeClustering(n_clusters=3,
affinity='euclidean',
linkage='complete')
labels = ac.fit_predict(X)
print('클러스터 레이블: ', labels)
6) Cluster distance measure
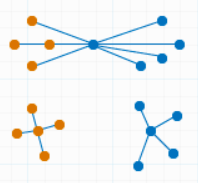
(1) single linkage
- 클러스터 쌍에서 가장 가까운 샘플 간 거리 계산하고 거리가 가장 작은 두 클러스터 병합
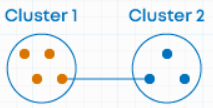
(2) complete linkage
- 클러스터 쌍에서 가장 먼 샘플 간 거리 계산하고 거리가 가장 작은 두 클러스터 병합

(3) average linkage
- 두 클러스터 간에 모든 샘플 사이의 평균 거리가 가장 작은 클러스터 쌍을 병합

(4) centroid method
- 두 클러스터의 centroids 간의 거리가 가장 작은 클러스터 쌍을 병합
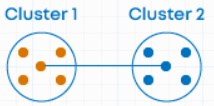
(5) Ward's method
- 클러스터 내 SSE가 가장 작게 증가하는 두 클러스터를 병합
$$Ward \: Distance = \sum_{i \in A\cup B}||x_{i}-m_{A\cup B}||^{2}-(\sum_{i\in A}||x_{i}-m_{A}||^{2}+\sum_{i\in B}||x_{i}-m_{B}||^{2})$$
K-Means Clustering (k-평균 군집화)
1) 개요
- 클러스터 개수를 사전에 지정해야 하는 단점이 있다.
- 클러스터가 중첩되지 않고 계층적이지 않다.
- 연속 데이터에서는 centroid, 범주 데이터에서는 medoid를 가진다.
2) 과정
(1) k개의 중심을 임의로 생성
(2) 생성된 중심을 기준으로 모든 관측치에 군집 할당
(3) 2에서 생성된 군집의 중심을 다시 계산
(4) 중심이 변하지 않거나 지정한 허용 오차나 최대 반복 회수에 도달할 때까지 위의 과정 반복
3) 단점
- 서로 다른 크기의 군집을 잘 찾아내지 못함
- 서로 다른 밀도의 군집을 잘 찾아내지 못함
- 지역적 패턴이 존재하는 군집을 판별하기 어려움

4) K-means ++
- 초기 centroid가 서로 멀리 떨어지도록 위치시킨다.
from sklearn.cluster import KMeans
km = KMeans(n_clusters = 3, init = 'k-means++', n_init = 10,
max_iter = 300, tol = 1e-04, random_state = 0)
y_km = km.fit_predict(X)DBSCAN (Density-Based Spatial Clustering of Applications with Noise)
1) 개요
- k-means처럼 원형 클러스터를 가정하지 않는다.
- 계층적인 방식으로 데이터셋을 나누지 않는다.
- 결과 평가 시, 차원 축소를 하여 클러스터를 시각화하고 레이블을 할당하여 결과 평가에 도움된다.
- 샘플이 조밀하게 모인 지역에 클러스터 레이블 할당
2) 조건
- 어떤 샘플의 반경 안에 있는 이웃 샘플이 지정된 개수 (MinPts) 이상이면 핵심 샘플이 된다.
- 반경 이내에 MinPts보다 이웃이 적지만 다른 핵심 샘플의 반경 안에 있으면 경계 샘플이 된다.
- 핵심, 경계 샘플이 모두 아닌 경우 잡음 샘플이 된다.
3) 과정
(1) 개별 핵심 샘플이나 핵심 샘플의 그룹을 클러스터로 만든다.
(2) 경계 샘플을 해당 핵심 샘플의 클러스터에 할당한다.
4) 코드 예시
from sklearn.cluster import DBSCAN
db = DBSCAN(eps = 0.2,
min_samples = 5,
metric = 'euclidean')
y_db = db.fit_predict(X)
plt.scatter(X[y_db == 0, 0],
X[y_db == 0, 1],
s = 40, c = 'lightblue',
marker = 'o', edgecolor = 'black',
label = 'cluster 1')
plt.scatter(X[y_db == 1, 0],
X[y_db == 1, 1],
s = 40, c = 'red',
marker = 's', edgecolor = 'black',
label = 'cluster 2')
plt.legend()
plt.tight_layout()
plt.show()
군집화 평가 지표
1) SSE (Sum of Squared Error)
- 중심 C_i와 관측치 x 거리 제곱의 합이 줄어드는 지점
$$SSE = \sum_{i=1}^{K}\sum_{x\in C_{i}}dist(x, C_{i})^{2}$$
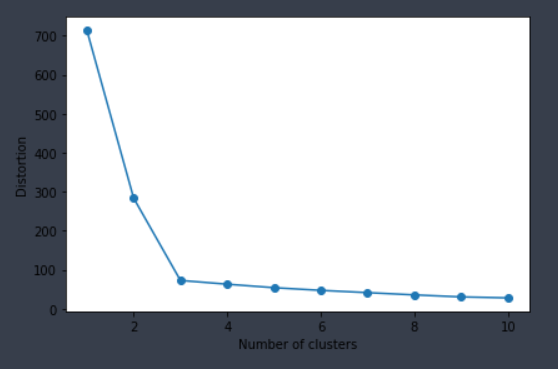
distortions = []
for i in range(1, 11):
km = KMeans(n_clusters = i,
init = 'k-means++',
n_init = 10,
max_iter = 300,
random_state = 0)
km.fit(X)
distortions.append(km.inertia_) '''inertia_ : SSE'''
plt.plot(range(1, 11), distortions, marker = 'o')
plt.xlabel('Number of clusters')
plt.ylabel('Distortion')
plt.tight_layout()
plt.show()
2) Silhouette analysis
- 클러스터 내 샘플들이 얼마나 조밀하게 모여 있는지 측정하는 그래프 도구
$$s_{i} = \frac{b_{i}-a_{i}}{max(b_{i}, a_{i})}$$
- bi : 샘플 xi와 가장 가까운 클러스터의 모든 샘플 간 평균 거리로 최근접 클러스터의 클러스터 분리도
- ai : 샘플 xi와 동일한 클러스터 내 모든 다른 포인트 사이의 거리를 평균하여 클러스터 응집력
- -1 <= si <= 1 : 1에 가까울수록 군집화가 잘된 것이다.
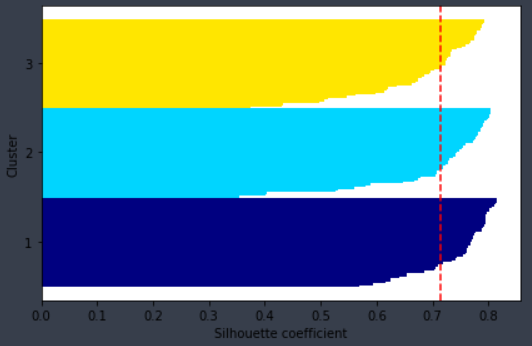
km = KMeans(n_clusters = 3,
init = 'k-means++',
n_init = 10,
max_iter = 300,
tol = 1e-04,
random_state = 0)
y_km = km.fit_predict(X)
import numpy as np
from matplotlib import cm
from sklearn.metrics import silhouette_samples
cluster_labels = np.unique(y_km)
n_clusters = cluster_labels.shape[0]
silhouette_vals = silhouette_samples(X, y_km, metric = 'euclidean')
y_ax_lower, y_ax_upper = 0, 0
yticks = []
for i, c in enumerate(cluster_labels):
c_silhouette_vals = silhouette_vals[y_km ==c]
c_silhouette_vals.sort()
y_ax_upper += len(c_silhouette_vals)
color = cm.jet(float(i) / n_clusters)
plt.barh(range(y_ax_lower, y_ax_upper),
c_silhouette_vals,
height = 1.0,
edgecolor = 'none',
color = color)
yticks.append((y_ax_lower + y_ax_upper) / 2.)
y_ax_lower += len(c_silhouette_vals)
silhouette_avg = np.mean(silhouette_vals)
plt.axvline(silhouette_avg, color = 'red', linestyle = '--')
plt.yticks(yticks, cluster_labels + 1)
plt.ylabel('Cluster')
plt.xlabel('Silhouette coefficient')
plt.tight_layout()
plt.show()참고
머신러닝 교과서 (길벗)
고려대 김성범 교수님 유튜브 www.youtube.com/channel/UCueLU1pCvFlM8Y8sth7a6RQ/videos
'AI > Machine Learning' 카테고리의 다른 글
| Logistic regression (0) | 2021.03.02 |
|---|---|
| Linear regression (0) | 2021.02.26 |
| Ensemble (Voting, Bagging, Boosting) (0) | 2021.02.24 |
| Model evaluation metrics (0) | 2021.02.23 |
| Validation (Cross-val, learning-curve, GridSearch) (0) | 2021.02.23 |
